تحلیل دادههای ژنومی یکی از بخشهای مهم بیوانفورماتیک و زیستشناسی محاسباتی است که با استفاده از ابزارها و روشهای پیشرفته به بررسی ساختار، توالی و عملکرد ژنوم موجودات مختلف میپردازد. این تحلیلها به محققان کمک میکند تا به درک عمیقتری از ژنتیک، فرگشت و بیماریها دست پیدا کنند.
نرمافزارها و ابزارهای تحلیل دادههای ژنومی
برای تحلیل دادههای ژنومی، ابزارهای متنوعی وجود دارد که هرکدام کاربرد خاص خود را دارند. در زیر برخی از مهمترین ابزارها معرفی میکنیم.
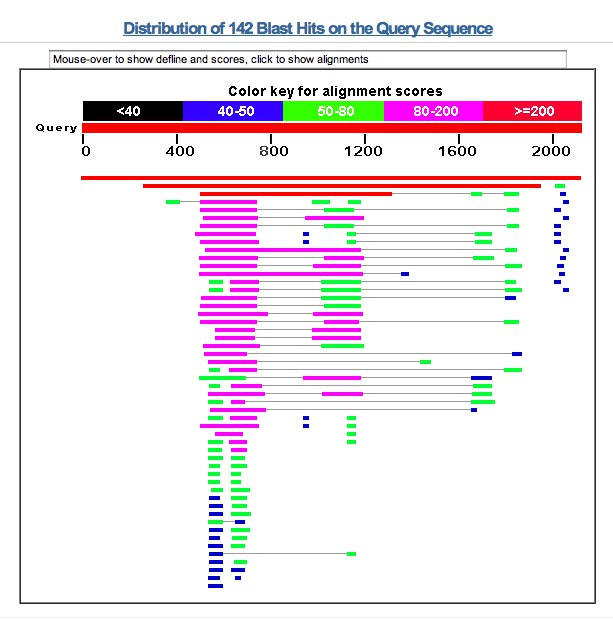
نرمافزار بلاست
بلاست(BLAST) یکی از رایجترین ابزارها برای جستجوی شباهت توالیها است. این نرمافزار با مقایسه توالی مورد نظر با پایگاهدادههای ژنتیکی، امکان شناسایی توالیهای مشابه و تعیین عملکرد احتمالی آنها را فراهم میکند. این نرمافزار میتواند تحلیل توالیهای DNA، RNA و پروتئین را انجام دهد.
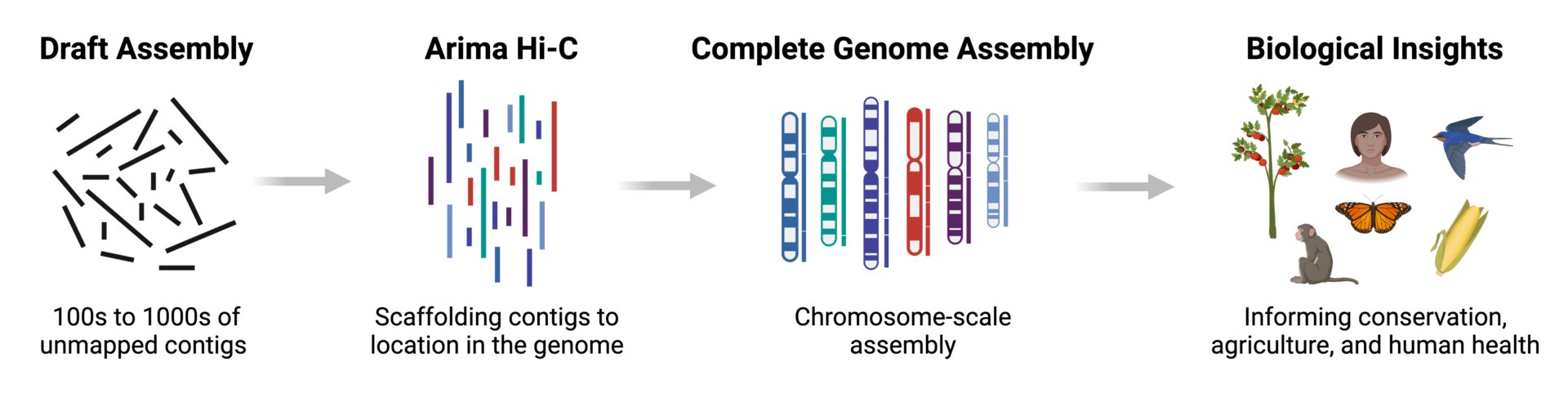
ابزار های سرهم بندی ژنوم
این ابزارها برای سرهمبندی توالیهای کوتاه DNA به یک ژنوم کامل استفاده میشوند. بعضی از این ابزارها عبارتند از SPAdes, Velvet و Canu.
ابزار GATK
ابزار پیشرفتهای برای شناسایی جهشها، تغییرات ساختاری و تغییرات تکنوکلئوتیدی (SNP) در دادههای ژنومی که در بررسی بیماریهای ژنتیکی بکار میآید.
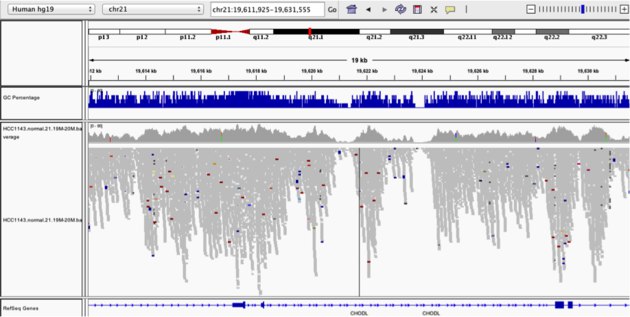
نرمافزار IGV
نرمافزاری برای مشاهده تعاملی دادههای ژنومی میباشد که در تجسم دادههای توالییابی و تعیین مکان جهشها کاربرد دارد.
روشهای بیوانفورماتیکی برای تحلیل دادهها
جستجوی همترازی توالیها
این روش برای پیدا کردن شباهتها و تفاوتهای توالیها استفاده میشود. دو نوع همترازی وجود دارد:
- همترازی محلی: تمرکز بر شباهت یک بخش خاص از توالی
- همترازی سراسری: مقایسه کل توالی
ابزارهای BLAST، ClustalW و MAFFT، ابزار های رایجی در این زمینه هستند.

سرهمبندی ژنوم(Genome Assembly)
هدف این روش، بازسازی ژنوم کامل از قطعات کوچک DNA است که توسط دستگاههای توالییابی تولید میشوند.
- روش De novo Assembly: بدون مرجع.
- روش Reference-based Assembly: با استفاده از ژنوم مرجع.
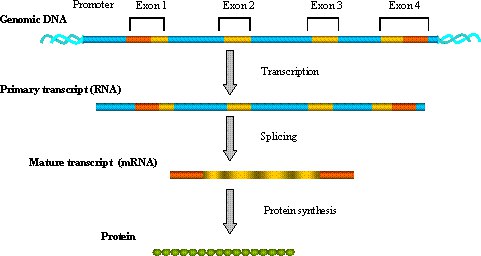
تجزیه و تحلیل بیان ژن (Gene Expression Analysis)
در این روش با استفاده از ابزازهای RNA-Seq، EdgeR، DESeq. برای بررسی سطح بیان ژنها در شرایط مختلف زیستی یا بیماریها استفاده میشود.
تجزیه و تحلیل جهشها (Variant Analysis)
تکنیکی برای شناسایی تغییرات ژن است. ابزارهای GATK، ANNOVAR، VEP در اینباره بسیار عملکرد خوبی دارند.
پیشبینی ساختار پروتئین و عملکرد ژنها
مدلسازی همولوژی (Homology Modeling)، Docking، شبیهسازی دینامیک مولکولی که همگی با استفاده ازابزارهای SWISS-MODEL، Phyre2، AutoDock انجام میشود.
منابع
- https://digitalworldbiology.com/products/blast-resources
- https://rnabio.org/module-02-alignment/0002/04/01/IGV/
- https://arimagenomics.com/applications/genome-assembly/
- https://www.ncbi.nlm.nih.gov/probe/docs/applexpression/#:~:text=Gene%20expression%20analysis,or%20in%20a%20specific%20cell.
- https://www.ebi.ac.uk/training/online/courses/human-genetic-variation-introduction/variant-identification-and-analysis/



